(通讯员 刘小磊)近日,赵书红教授团队在Nature子刊《Communications Biology》杂志上在线发表了题为“A gene prioritization method based on a swine multi-omics knowledgebase and a deep learning model”的研究成果。本研究发布了国际上首个猪整合组学知识库ISwine,创建了一个基于卷积神经网络模型和多组学信息的候选基因评分推荐系统,打通了从GWAS结果的显著标记到候选基因推荐的“最后一公里”。
单一组学的分析(如GWAS)往往止步于标记和表型间的“相关”,难以揭示“因果”,中心法则告诉我们遗传信息从DNA转录到RNA,再翻译生成各种蛋白质,行使特定的生物功能,一个完整的生物过程需要多个组学的参与。近年来,猪的多组学信息呈超指数型增长,如何解读海量异质性的多组学数据,整合来自不同研究的多组学信息来解析遗传变异与重要经济性状间的关系面临着极大的挑战。
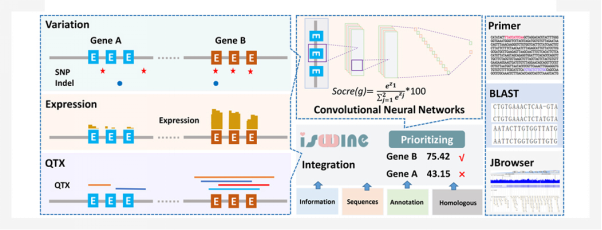
为解决上述问题,团队收集了公共数据库中近乎所有的猪基因组数据、转录组数据以及性状相关的文献组数据。通过清洗、分析、以及结构化等过程,将这些数据以基因组变异数据库、基因表达数据库以及QTX数据库的形式收录到ISwine中。其中基因表达数据库是猪中第一个基于转录组数据的表达谱数据库,基因变异数据库是猪中最大的变异信息数据库, NCBI在2018年停止了对猪dbSNP数据库的更新,ISwine将为猪遗传育种研究人员提供丰富的基因组变异信息和完备的单倍型信息。另外,ISwine根据不同组学特点提供了用户界面友好的浏览、检索、可视化、交互和下载模块,用户可以节约大量的分析时间和费用,方便快捷的利用这些海量的组学信息,例如直接输入GWAS结果或候选基因列表,ISwine会基于卷积神经网络模型和多组学信息针对目标性状推荐“高分”候选基因。该策略可拓展应用到其他物种,为多组学信息在遗传和育种中的应用提供了新思路。
我校和武汉理工大学计算机科学与技术学院联合培养的博士后付玉华为论文第一作者。我校赵书红教授、刘小磊副教授和武汉理工大学袁晓辉教授为文章共同通讯作者,上述研究工作得到了国家自然科学基金等项目的资助。
ISwine知识库:http://iswine.iomics.pro
ISwine使用示例:http://iswine.iomics.pro/pig-iqgs/commonView?viewName=Tutorial
原文链接:https://www.nature.com/articles/s42003-020-01233-4

