(通讯员 谢胜松)近日,米兰(中国)动物遗传育种团队在Nature子刊《Nature Communications》杂志上在线发表了题为“CRISPR screening of porcine sgRNA library identifies host factors associated with Japanese encephalitis virus replication”的研究成果。该研究首次构建了基于猪全基因组CRISPR/Cas9敲除文库技术的高通量功能基因筛选平台,并运用该技术筛选和鉴定出参与乙型脑炎病毒复制的必需宿主基因。这一研究对猪抗病育种及具有育种价值的关键功能基因发掘具有重要参考价值。
随着家猪基因组测序技术日趋完善,以及全基因组选择在猪育种中逐步应用,家猪基因组学的研究快速进入功能基因组时代。借助CRISPR/Cas9基因编辑技术,国内外已经构建出多种基因编辑猪模型。然而,具有重大育种价值的可编辑基因和靶点仍然缺乏,制约了猪基因编辑育种研究工作进程,基因编辑技术的发展为从全基因组水平筛选重要功能基因提供了契机。基于此,以家猪为对象,该研究团队利用自主开发的sgRNA文库设计软件,针对猪的几乎全部蛋白编码基因,长链非编码RNA和微小RNA,设计和构建了一个高质量sgRNA表达载体文库与突变细胞文库(PigGeCKO, v1.0,图1)。随后,该研究团队利用猪的全基因组CRISPR敲除文库技术,成功实现了高通量筛选和鉴定参与乙型脑炎病毒复制的关键宿主因子。
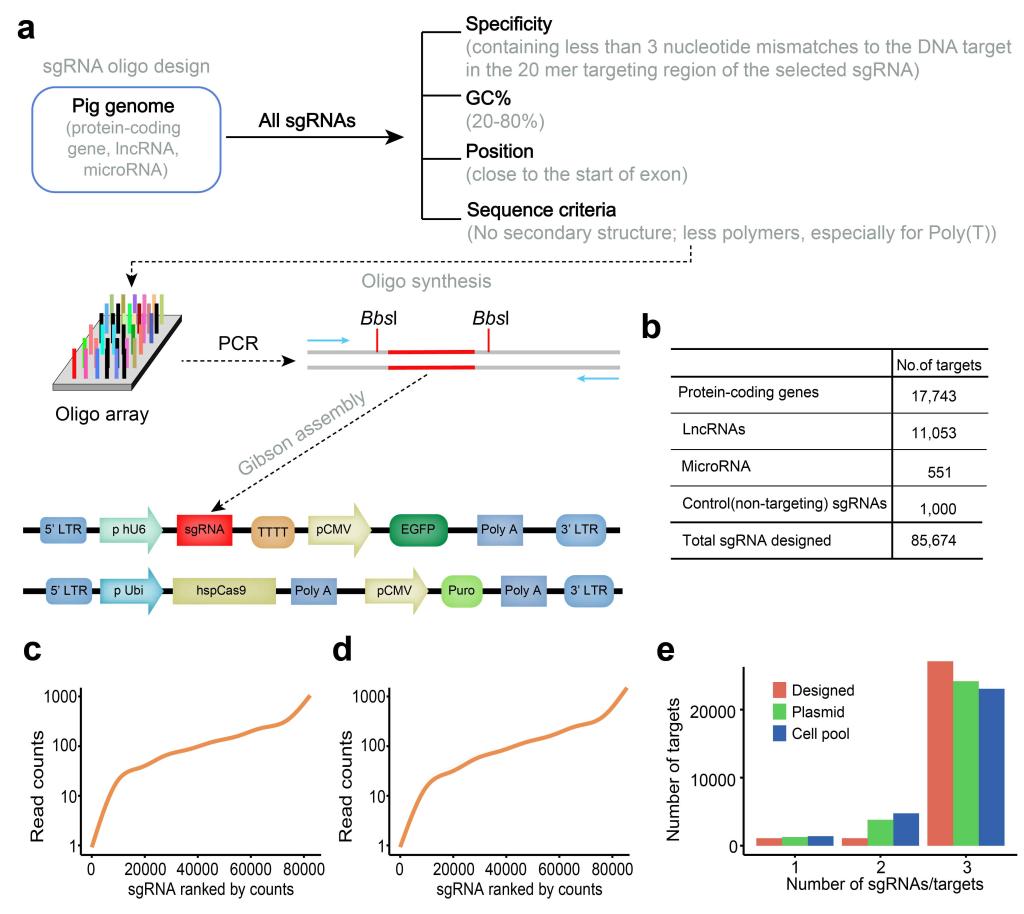
图1.猪全基因组CRISPR敲除文库(PigGeCKO)设计和构建
基于全基因组CRISPR文库设计构建关键功能基因高通量筛选平台,对于功能基因组研究和品种改良具有重要产业价值,将大力推动家猪功能基因鉴定进入精准、高通量研究的新阶段,为基因功能鉴定和育种提供新思路和新策略。近年来,该团队在sgRNA设计软件开发(biootools.com)、高效准确评估sgRNA活性与脱靶效应、基于CRISPR筛选文库的功能基因挖掘及基因编辑猪育种新材料创制等方面取得系统性研究成果,为猪功能基因组与育种研究增添了新利器。
米兰(中国)博士赵长志、硕士刘海龙和肖天贺为文章共同第一作者,赵书红教授、谢胜松副教授为文章共同通讯作者,上述研究工作得到了转基因生物新品种培育重大专项、国家自然科学基金等项目资助。
原文链接:https://www.nature.com/articles/s41467-020-18936-1

